- 产品详情
- 产品参数
- 产品评论
蛋白质主要由碳、氢、氧、氮等化学元素组成,是一类重要的生物大分子。蛋白质的功能由蛋白质的三维结构决定。蛋白质三维结构绘图,可以直观地展示蛋白质三维功能结构,广泛应用于单核苷酸突变功能分析、药物蛋白分子相互作用分析等研究领域。
基本原理
蛋白质三维结构绘图主要分为蛋白质三维结构预测以及对结构进行可视化两步。
蛋白质三维结构预测是基于蛋白质中氨基酸序列预测蛋白质折叠结构的步骤,最常用的预测方法为同源建模,同源建模的原理是序列相似的蛋白质具有相似的蛋白质结构,要推测一个未知结构蛋白的三维结构,只需要找到与之序列高度相似的已知结构模板。在无法进行同源建模(找不到模型)的情况下,还有折叠识别及从头建模法,但是计算量大运行缓慢且建模准确度不如同源建模。
获得蛋白质三维结构预测的pbd文件后还需要通过分子三维结构软件绘制可视化的三维图,并分析特殊位点(分子对接或突变位点分析),常用的有pymol和DeepView等。
数据要求
目标蛋白的氨基酸序列或者编码蛋白的基因序列,突变数据等。
下游分析
突变位点靶向药物分析等。
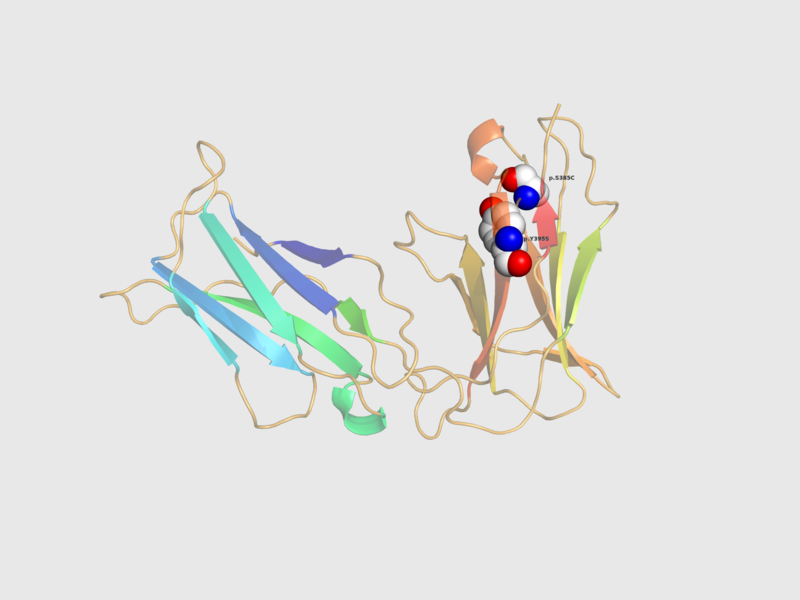
图形示例:
1、LILRA1三维结构1

2、LILRA1三维结构2

应用示例:
文献1:Clinicalutility of genetic variants of glutamate carboxypeptidase II in predictingbreast cancer and prostate cancer risk.(于2015年12月发表在cancer genetics,影响因子2.183)
GCPII基因突变对乳腺癌及前列腺癌预后的预测研究
本文基于GCPII基因的突变位点将乳腺癌及前列腺癌患者分为8种预后相关的亚型。作者绘制蛋白三维结构图展示了不同GCPII基因突变位点对蛋白质氨基酸序列的影响,及改变的氨基酸在三维结构上的位置。

并基于蛋白质三维结构进行了药物结合分析。

文献2:Acomputational approach to determine susceptibility to cancer by evaluating thedeleterious effect of nsSNP in XRCC1 gene on binding interaction of XRCC1protein with ligase III(于2016年1月发表在Gene,影响因子2.638)
XRCC1基因的nsSNP对XRCC1-LIG3相互作用及癌症易感性的影响分析
作者使用SIFT, PolyPhen, PANTHER, PhD-SNP, nsSNPAnalyzer等软件分析了XRCC1各种单核苷酸突变的有害性程度,找出了有害性最强的rs2307166。文中绘制蛋白质三维结构分析了单核苷酸突变对蛋白质结构和功能的影响,并进行分子对接分析了rs2307166对XRCC1-LIG3相互作用的影响。