- 产品详情
- 产品参数
- 产品评论
RNAseqChIP
根据RNA-seq表达谱分析得到的结果,绘制对应基因启动子区的ChIP-seq信号,观察转录因子对基因的调控影响。一般可应用场景:测了RNA-seq和ChIP-seq,结合转录因子结合情况分析基因表达;只测了RNA-seq,补充相关ChIP-seq公共数据。
基本原理:
染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP)也称结合位点分析法,是一种研究蛋白质与染色质结合情况的方法。将ChIP与第二代测序技术相结合的ChIP-Seq,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的DNA区段。
转录组测序RNA-seq,获取的转录组基因表达情况,结合ChIP-seq数据,可以从更宏观的角度分析转录因子调控的对基因表达的影响。
数据要求:
基因列表,ChIP-seq数据。
下游分析:
对得到的聚类基因进行后续分析:
1.不同簇基因的功能富集
2.不同簇基因与性状的相关性分析
图形示例:
1.ChIP-seq聚类热图

图例:左侧为不同类基因类名,下方为基因启动子区结合位点3kb区域。
应用示例:
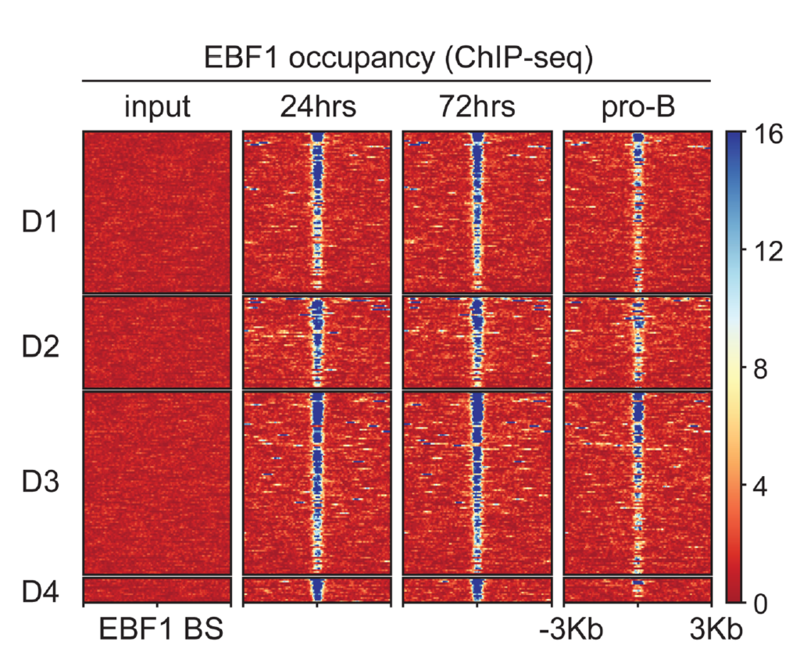
文献1:Dynamic EBF1occupancy directs sequential epigenetic and transcriptional events in B-cellprogramming(于2018年1月发表在Genes Dev.,影响因子3.331)
EBF1动态占据在B细胞中对序列表观遗传和转录过程的影响

该文献对ChIP-seq数据根据RNA-seq表达情况进行排列展示,展示了EBF1靶向结合位点与EBF1的结合情况及染色质开放情况。
文献2:The histonedemethylase KDM3A regulates the transcriptional program of the androgenreceptor in prostate cancer cells(于2017年5月发表在Oncotarget. ,影响因子3.046)
组蛋白去甲基化酶KDM3A调节前列腺癌细胞中雄激素受体的转录过程

该文献将对敲除KDM3A后KDM3A的ChIP-Seq信号情况、H3K9me1、H3K9me2和雄激素受体绘制了热图,观察KDM3A敲除前后甲基化和雄激素受体作用变化。