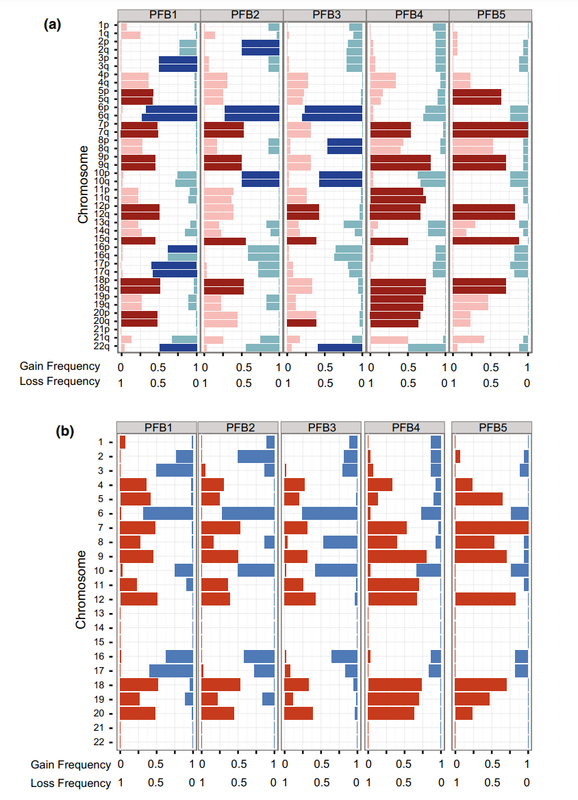
不同分组的全基因组拷贝数变化的比较
收藏
¥0.00
收藏
- 产品详情
- 产品参数
- 产品评论
最初目的:不同分组的拷贝数变异在染色体水平和染色体臂水平的展示和比较。
应用:不同分组的全基因组拷贝数变化的比较,展示genome-wideDNA copy-number profiles。不同染色体臂的变异与临床表型息息相关。
输入数据格式:一个表征每个样本的染色体变异(gain, balance, loss)的数值矩阵和样本分组信息。或者拷贝数的原始结果,可处理成所需矩阵。
参考文献:
参考文献思路:
本文计算出病人的拷贝数变异情况后,按照之前病人的分组比较了不同分组的染色体变异的异同,找到特定的染色体变异模式。确定了各组的特征,如lmonosomy 2 in PFB2, monosomy 8 in PFB3, monosomy 3 in PFB1, andgain of 1q in PFB1.

我们出图效果:
1)染色体臂水平的差异

2)染色体水平的差异比较

服务时间
1个工作日