- 产品详情
- 产品参数
- 产品评论
 | ——ssGSEA分析代码 |
ssGSEA(single sample GSEA)主要针对单样本无法做GSEA而提出的一种实现方法,原理上与GSEA是类似的。ssGSEA根据表达谱文件计算每个基因的rank值,再进行后续的统计分析。通过这个方法,我们可以得到每个样本的免疫细胞或者免疫功能,免疫通路的活性,然后根据免疫活性进行分组。
ssGSEA量化免疫细胞浸润最大的一个优点就是自己可以定制量化免疫浸润细胞种类。目前公认并且用的最多的免疫细胞marker就是2013年发表在Immunity上的SpatiotemporalDynamicsof IntratumoralImmuneCellsReveal the Immune Landscape in Human Cancer 所提供的免疫细胞marker genes(Table S1),能提取到24种免疫细胞信息。
基本原理
对于一个基因表达矩阵,ssGSEA首先对样本的所有基因的表达水平进行排序获得其在所有基因中的秩次rank。然后对于输入的基因集,从基因集中寻找表达数据里存在的基因并计数,并将这些基因的表达水平求和。接着基于上述求值,计算通路中每个基因的富集分数,并进一步打乱基因顺序重新计算富集分数,重复一千次,最后根据基因富集分数的分布计算p值整合基因集最终富集分数。
数据要求
1、特定感兴趣的基因集(通常为免疫细胞表面marker genes),列出基因集中基因
2、基因表达矩阵,为经过log2标准化的芯片数据或者RNA-seq count数数据(基因名形式与基因集对应)
下游分析
免疫细胞浸润分数相关性(corralation)分析
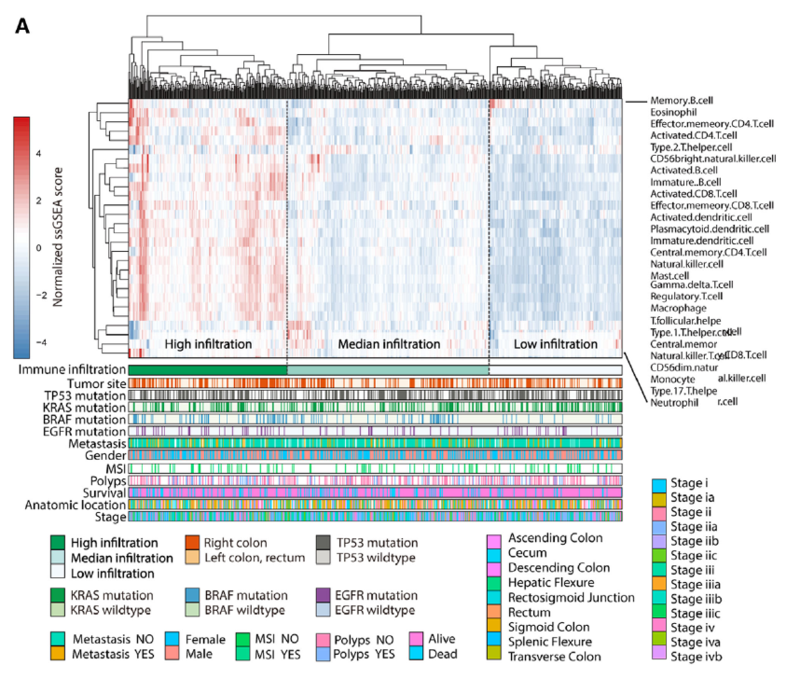
图形示例:

图注
上方热图部分为免疫细胞浸润分数,根据富集划分为高浸润和低浸润两组。下方热图为每个样本的其他指标,具体参考左边图例。
应用示例:
1、Pan-cancerImmunogenomic Analyses Reveal Genotype-Immunophenotype Relationships andPredictors of Response to Checkpoint Blockade.(于2017年1月发表在Cell Rep. ,影响因子7.815)
泛癌免疫基因组分析揭示基因型与免疫表型的相关性以及
对免疫功能调节药物反应的预测
文章研究了20种癌症的基因组数据,并对基因组学数据进行了免疫相关分析。文中利用ssGSEA识别了肿瘤微环境中过表达的免疫细胞的种类。
2、Localmutational diversity drives intratumoral immune heterogeneity in non-small celllung cancer(于2018年12月发表在Nat Commun. ,影响因子11.878)
非小球肺癌中局部突变多样性驱动的瘤内免疫异质研究
文中结合全基因组测序、转录分析和T细胞库分析研究了免疫微环境的空间异质。其中利用ssGSEA研究了肿瘤微环境的免疫细胞浸润。